重庆邮电大学
fluid-attenuated inversion recovery (FLAIR)
T1-weighted (T1)
contrast-enhanced T1-weighted(T1ce)
T2-weighted (T2)
摘要
Brain tumor segmentation is currently of a priori guiding significance in medical research and clinical diagnosis. Brain tumor segmentation techniques can accurately partition different tumor areas on multi-modality images captured by magnetic resonance imaging (MRI). Due to the unpredictable pathological process of brain tumor generation and growth, brain tumor images often show irregular shapes and uneven internal gray levels. Existing neural network-based segmentation methods with an encoding/decoding structure can perform image segmentation to some extent. However, they ignore issues such as differences in multi-modality information, loss of spatial information, and under-utilization of boundary information, thereby limiting the further improvement of segmentation accuracy. This paper proposes a multimodal spatial information enhancement and boundary shape correction method consisting of a modality information extraction (MIE) module, a spatial information enhancement (SIE) module, and a boundary shape correction (BSC) module. The above three modules act on the input, backbone, and loss functions of deep convolutional networks (DCNN), respectively, and compose an end-to-end 3D brain tumor segmentation model. The three proposed modules can solve the low utilization rate of effective modality information, the insufficient spatial information acquisition ability, and the improper segmentation of key boundary positions can be solved. The proposed method was validated on BraTS2017, 2018, and 2019 datasets. Comparative experimental results confirmed the effectiveness and superiority of the proposed method over state-of-the-art segmentation methods.
翻译
脑肿瘤分割在医学研究和临床诊断中具有优先指导意义。脑肿瘤分割技术可以准确地在磁共振成像(MRI)捕获的多模态图像上划分不同的肿瘤区域。由于脑肿瘤生成和生长的病理过程不可预测,脑肿瘤图像常常表现出不规则形状和内部灰度不均匀。现有基于神经网络的分割方法采用编码/解码结构,能够在一定程度上进行图像分割。然而,它们忽视了多模态信息的差异、空间信息的丢失和边界信息的利用不足等问题,从而限制了分割精度的进一步提高。本文提出了一种多模态空间信息增强和边界形状校正方法,该方法由模态信息提取(MIE)模块、空间信息增强(SIE)模块和边界形状校正(BSC)模块组成。上述三个模块分别作用于深度卷积网络(DCNN)的输入、骨干网络和损失函数,并构成一个端到端的三维脑肿瘤分割模型。这三个提出的模块可以解决有效模态信息利用率低、空间信息获取能力不足以及关键边界位置分割不当的问题。所提出的方法在BraTS2017、2018和2019数据集上进行了验证。比较实验结果证实了该方法相较于最先进的分割方法的有效性和优越性。
研究背景
本文聚焦于脑肿瘤磁共振成像(MRI)分割,其研究背景主要源于脑肿瘤的高风险及精准分割的重要性,现有方法存在的不足,具体如下:
- 脑肿瘤的危害与精准分割的重要性:脑肿瘤是脑组织癌变引发的异常细胞生长,会导致头痛、认知问题等症状,严重时危及生命。准确识别脑肿瘤位置和形态对临床至关重要,而MRI是诊断和治疗脑肿瘤的重要工具,可提供软组织解剖结构的高分辨率图像。
- 现有方法的不足:当前基于神经网络的分割方法虽有一定效果,但存在诸多局限性。一方面,序列图像结构边界模糊、特征不清晰,且现有方法忽视多模态信息差异,仅叠加多模态MRI扫描结果,导致有价值的多模态信息利用不充分;另一方面,基于卷积神经网络(CNNs)的方法主要捕捉局部信息,难以有效保留表征三维空间全局上下文关系的空间信息,同时忽略边界信息的利用,降低了分割准确性。现有方法尚未能很好地解决这些问题,因此本文提出多模态空间信息增强和边界形状校正方法,以提高脑肿瘤分割的精度。
研究现状
- 医学图像分割方法:传统方法基于机器学习,如阈值、区域、边缘分割法,但易受噪声影响。深度学习方法通过神经网络训练分类像素,能解决部分问题,综合性能佳。
- 维度处理:包括2D和3D处理方法。2D先切片再拼接,经典网络如FCN、UNet最初用此方法;3D能提供更全面空间信息和立体效果,典型网络有V - Net。
- 多模态脑肿瘤图像分割:BraTS挑战赛推动了相关研究,提出多种创新网络结构,但对多模态和边界稳定性研究不足
提出的模型
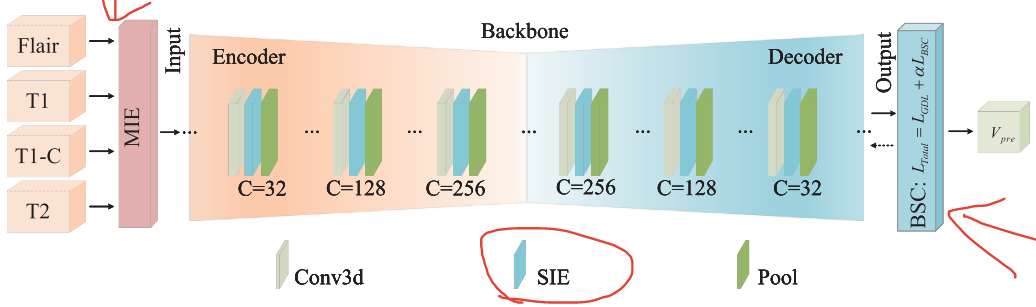
文章提出了一种用于脑肿瘤分割的多模态空间信息增强和边界形状校正方法,该方法由模态信息提取(MIE)模块、空间信息增强(SIE)模块和边界形状校正(BSC)模块组成,形成了一个端到端的3D脑肿瘤分割模型。
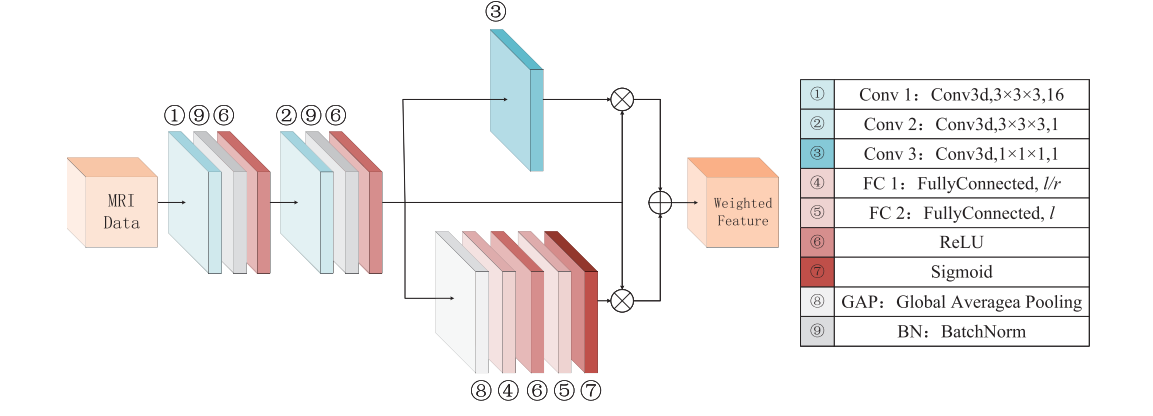
模态信息提取(MIE)模块
- 作用:解决多模态信息差异问题,通过注意力机制在网络训练期间为关键模态信息分配更高权重,增强多模态信息的融合效果。
- 工作流程:首先将所有模态的三维数据在数据通道维度上合并转换为四维数据,利用两组3D卷积、正则化和激活操作提取各模态的图像特征;然后引入sc - SENet结构,对输出特征数据进行空间和通道重要性加权,得到加权特征作为骨干分割网络的输入数据。
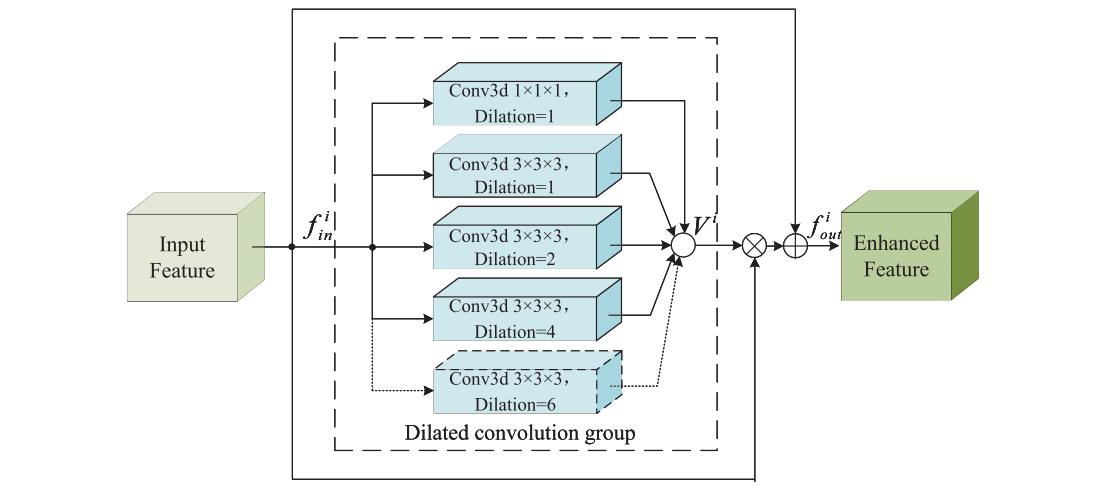
空间信息增强(SIE)模块
- 作用:解决深度卷积分割网络中空间信息丢失的问题,通过多尺度扩张卷积组扩大空间感受野,增强特征图中的空间信息,使网络有效探索3D图像数据各层之间的全局关系。
- 工作流程:该模块位于基线编码/解码过程的卷积层之后、池化层之前,接收卷积层输出的特征图像作为输入,通过扩张卷积组生成$V_i$,再将$V_i$融合到原始特征图中,增强原始输入特征图像的空间信息。输入和输出可表示为$f_{i_{out}}=(f_{i_{in}}\otimes V_i)\oplus f_{i_{in}}$。
边界形状校正(BSC)模块
- 作用:解决边界信息利用不足的问题,通过空间关键边界点选择算法和边界校正损失函数,提高预测图像在关键边界位置的分割性能。
- 空间关键边界点选择算法:在不规则形状的三维图像中选择一组空间关键边界点,有效恢复肿瘤形状。具体步骤包括边界表面提取、边界点选择、通过迭代选择最佳点集。
- 边界校正损失函数:使用空间关键边界点选择算法生成关键边界点,将关键边界点图像与预测图像的重叠关系设置为损失函数,通过梯度反向传播最大化两类图像之间的相关性,实现边界的不断修正。
该模型通过上述三个模块分别对输入、骨干和损失函数进行改进,进一步提高了脑肿瘤分析的分割性能。实验在BraTS2017、2018和2019数据集上进行,对比实验结果证实了该方法相对于现有先进分割方法的有效性和优越性。此外,该模型在细胞分割和皮肤病变分割等其他医学图像分割问题上也表现出良好的适应性。
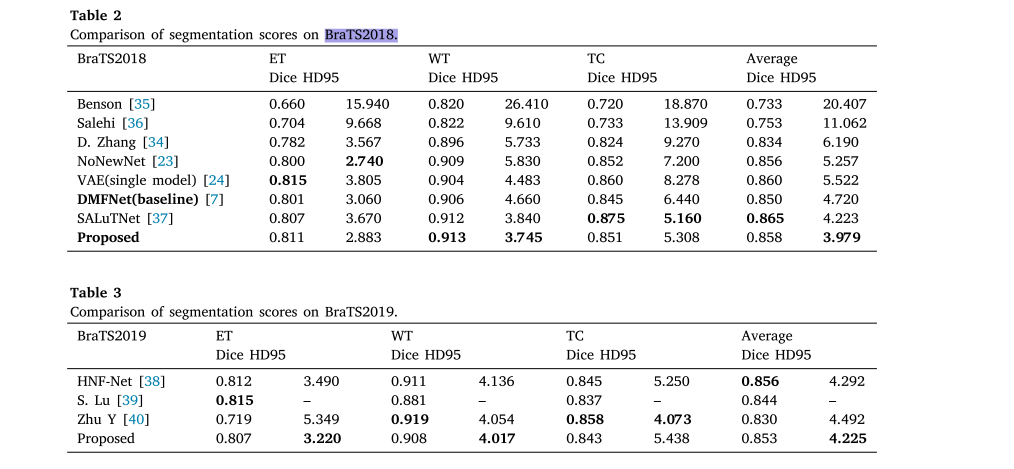
实验(Compared with SOTA)
实验准备:使用Pytorch程序框架实现网络,训练时使用8块TP100 GPU,采用Adam作为优化器,优化器动量为0.9,初始学习率为1×10⁻³,权重衰减为1×10⁻⁵,批量大小为8。对模型进行L2范数正则化,加权衰减率为1×10⁻⁵。从三个分割区域(WT、TC、ET)分别选择一定数量的点,迭代次数t为100,BSC模块中的R分别设置为2(WT)、1(TC)和1(ET)。广义Dice损失和定义的边界校正损失共同训练网络,至少需要500个训练周期,最短训练时间为12小时。
数据集:训练和测试数据集来自BraTS2017、BraTS2018和BraTS2019,这些数据集用于医学图像分割竞赛。每个病例有四种模态的3D体积数据,可直接由所提网络处理。采用Dice指数和Hausdorff95距离评估分割的准确性和性能。
实现细节
预处理:对原始数据进行标准化和归一化,使用数据增强技术,包括随机裁剪、随机镜像翻转、随机旋转和随机强度变换等,以减少GPU工作量、提高训练速度和增加训练数据。
测试阶段:将MRI数据扩展后作为网络输入,采用测试时间增强(TTA)方法进行数据增强,计算多个版本的平均输出作为最终结果。在ET区域分割时设置阈值过滤噪声。
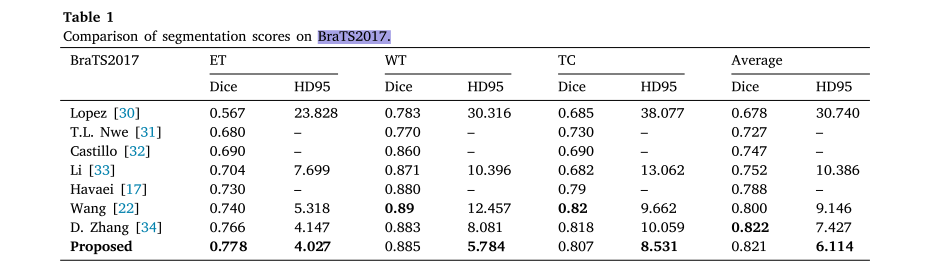
对比实验:将所提网络模型与多个先进的分割网络模型在BraTS2017、BraTS2018和BraTS2019数据集上进行对比实验。结果表明,所提方法在多个分割区域获得了更好的指标分数,尤其在肿瘤区域有显著提升。与先进的2D分割网络相比,所提的3D分割方法在客观评估指标上表现更优。
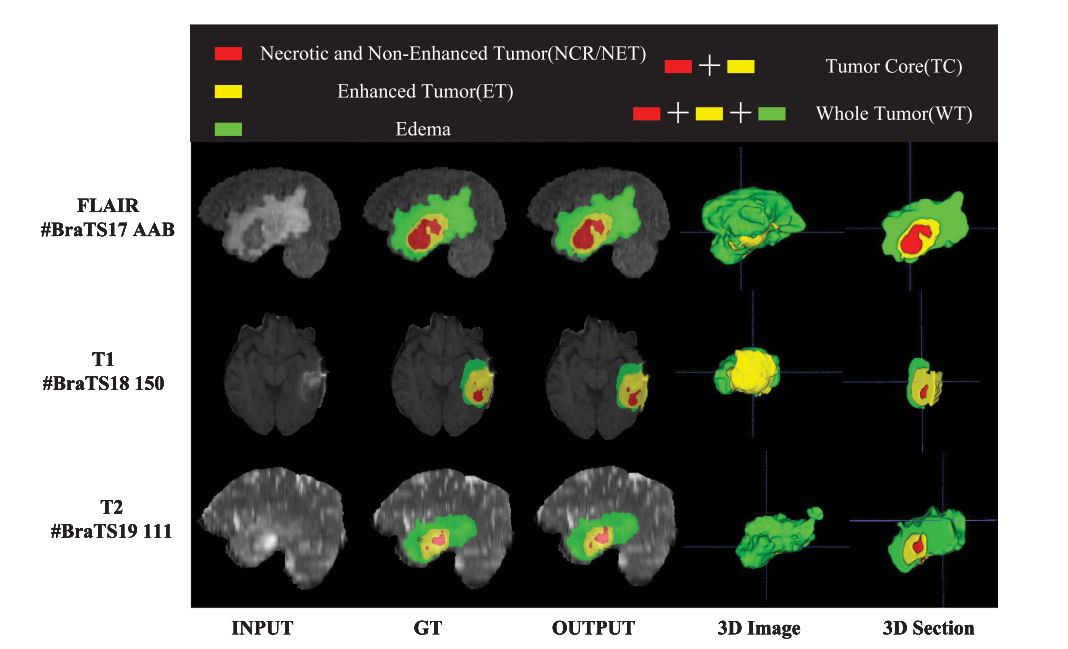
实验(Ablation Experiments)
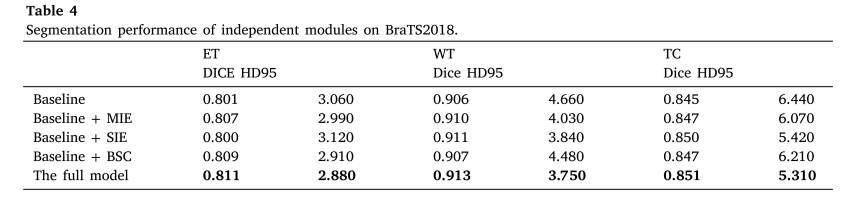
在相同基线(DMFNet)上逐个添加或减去模块进行消融实验。结果显示,三个功能模块在各分割区域的性能均有提升,不同模块在不同分割区域的贡献不同。所提的BSC损失函数能有效提高三个分割区域的分割性能。此外,添加模块后模型在可接受的模型大小和计算成本增加的情况下取得了最佳性能。
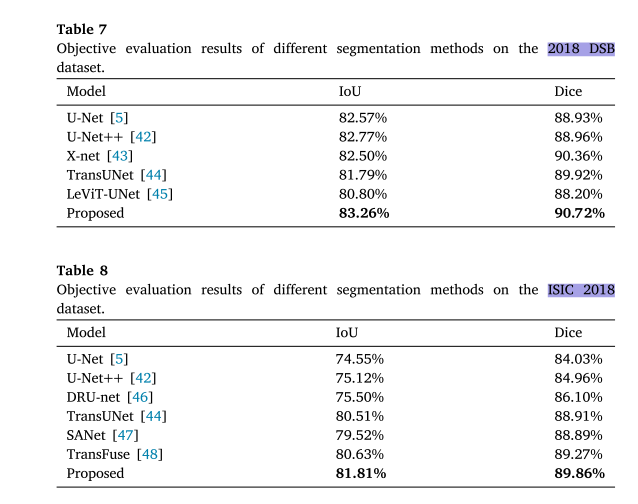
扩展到其他医学图像分割问题
选择细胞分割和皮肤病变分割两个医学图像分割问题进行测试,采用两个公开数据集。实验结果表明,所提模型在这两个问题上具有较好的适应性和竞争力。
结论
作者提出由MIE、SIE和BSC三个功能模块构成的新型结构框架,以解决脑肿瘤分割中的多模态、空间信息丢失和关键边界位置问题。研究结果表明:
- 各模块有效,该框架在脑肿瘤分割上优于现有方法。
- 该模型对其他医学图像分割问题(如细胞和皮肤病变分割)有良好适应性。
- 模型大小和计算成本在实际应用中总体可接受。 未来,作者将从性能和计算效率方面进一步优化分割模型,设计更有效架构和训练策略,并将框架扩展到更多医学图像分割问题,提升其在医学成像领域的适用性和影响力。



